2015-2025 között fejlesztett molekuláris genetikai módszerek
Az előző bejegyzésben lajstromozott technikákhoz képest számos területen történt jelentős fejlődés:
- a) célzottan, pontosan, sejttípusokat megcélozva lehet géneket változtatni, javítani, akár nagy méretű transzgéneket bevinni;
- b) külső behatással, fénnyel, vegyületekkel lehet géneket, receptorokat vagy ioncsatornákat időlegesen vagy tartósan ki-be kapcsolni;
- c) az „omics” technikák lehetővé teszik teljes rendszerek összefüggéseinek megértését;
- d) térbeli térképezési technikákkal teljes aktiválódó gén és fehérje kifejeződés-térképek készíthetők;
- e) a sejtvonalak és agyterületek fejlődését tudjuk befolyásolni;
- f) a nukleinsav és fehérje kölcsönhatások vizsgálatával a genetikai szabályozó hálózatok deríthetők fel;
- g) a genetikailag kódolt optikai érzékelő és ingerlő fehérjék lehetőségei jelentősen fejlődtek (erről már írtunk és még fogunk írni korábban)és végezetül
- h) az extrém multiomics, ahol számos nagy áteresztő képességű módszert kombinálnak teljes szabályozórendszerek vizsgálatára.
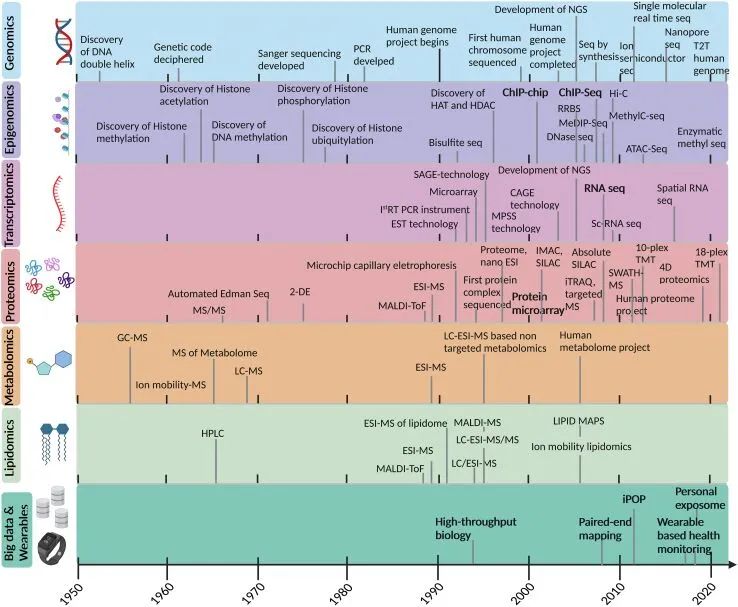
Azaz, képesek vagyunk hatalmas mennyiségű adatot gyűjteni 3D-ben, egész szervezetek, agyak, funkcionális állapotáról, szabályozó rendszereiről vagy megbetegedések esetén a változásokról. Színes, szagos, multimédiás vizsgálatok lettek elérhetők. Ezekhez persze komoly adatfeldolgozó háttér is kell, megfelelő softwarek és adatanalízis módszerek.
És akkor a meglehetősen hosszú lista, melyből a legelterjedtebb módszereket részletesen is bemutatjuk majd az elkövetkező blogbejegyzésekben.
 III. Modern genetikai szerkesztés és génbeviteli technikák: lehetővé teszik genetikai információ célzott bejuttatását sejtekbe, egyedekbe, pontos genetikai beavatkozásokat tesznek lehetővé, pl. mutációk kijavítását vagy betegségmodellek elkészítését.
III. Modern genetikai szerkesztés és génbeviteli technikák: lehetővé teszik genetikai információ célzott bejuttatását sejtekbe, egyedekbe, pontos genetikai beavatkozásokat tesznek lehetővé, pl. mutációk kijavítását vagy betegségmodellek elkészítését.
- 10. CRISPR-Cas9 / Cas12 / Cas13 rendszerek: génkiütés, pontmutáció, javítás
betegségmodellek létrehozása, in vivo génterápia alapja - 11. Base editorok (A→G, C→T átírás): DNS kettős-törés nélkül precíz pontmutációk beépítése, idegrendszeri betegségek modellezése
- 12. Prime editing: „molekuláris szövegszerkesztő”, akár több bázis cseréje / beszúrása / törlése is lehetséges, sejttoxicitás alacsony
- 13. Cre-loxP, Flp-FRT rekombinációs rendszerek: feltételes (conditional) kiütés, sejttípus-, életkor- vagy agyterület-specifikus genetikai módosítás, neurobiológia egyik alapeszköze
- 14. AAV, lentivírus, rabies vírusvektorok: génbevitel célzott sejtekbe
transz-szinaptikus tracerek (RV, HSV), optogenetikai fehérjék expressziója - 15. Transzpozáz alapú génbevitel (Sleeping Beauty, PiggyBac): stabil integráció, nagy méretű transzgén egyszerű bevitele
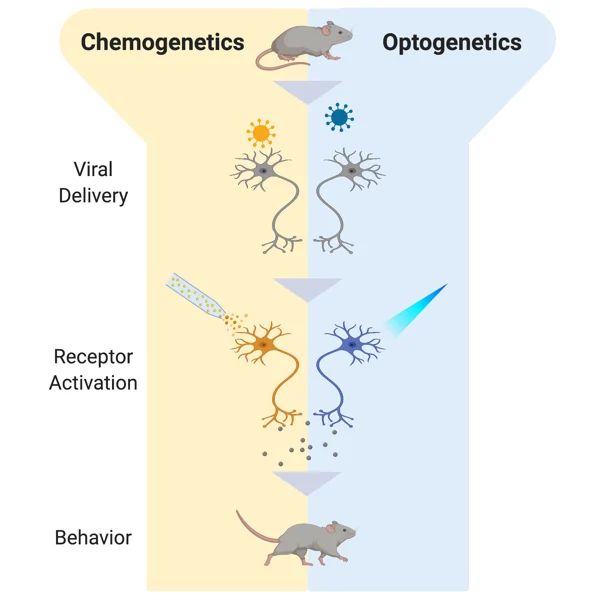 IV. Sejttípus- és környezet-specifikus génmanipuláció: gének kifejeződésének vagy fehérjék működésének célzott, időzített ki vagy bekapcsolását teszik lehetővé. Az agy embriológiai folyamatai és hálózatok működés vizsgálható ezekkel.
IV. Sejttípus- és környezet-specifikus génmanipuláció: gének kifejeződésének vagy fehérjék működésének célzott, időzített ki vagy bekapcsolását teszik lehetővé. Az agy embriológiai folyamatai és hálózatok működés vizsgálható ezekkel.
- 16. DREADD rendszer (chemogenetika): Gi, Gq, Gs jelátviteli utak mesterséges aktiválása, viselkedési és hálózatszintű vizsgálatok
- 17. Optogenetika (Channelrhodopsin, Halorhodopsin, Chrimson, stb.)
fényvezérelt aktiváció/gátlás: neuronhálózatok dinamikájának feltárása - 18. Tet-On/Tet-Off rendszerek: időzített génexpresszió, krónikus vs. akut hatások elkülönítése
V. Nagy áteresztőképességű molekuláris profilalkotás (omics): Ezekkel a módszerekkel sejtek vagy agyterületek teljes RNS-, fehérje-, transzkripciós faktor- kifejeződését, epigenetikus-, metabolikus- állapotát lehet meghatározni, gyakran térképszerű, térbeli lokalizációval. Hihetetlenül felgyorsítják a sejttípusok felfedezését és lokalizációját valamint funkcionális és fejlődési sajátságaik felderítését. Lehetővé teszik a fejlődés során a génkifejeződés és genetikai szabályozás mintázatainak felderítését. Nem megcélzott dolgokra kérdeznek rá, hanem mindent azonosítanak és a mérést követő elemzés során bukkannak fel fontos új összefüggések, mechanizmusok.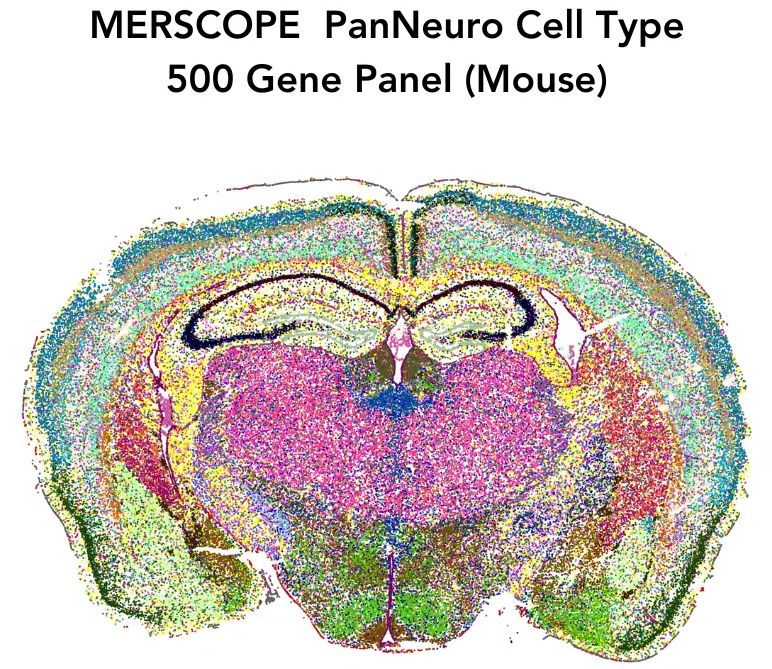
- 19. Bulk RNA-seq: agyterületek átlagos génexpressziója, fejlődési és betegségi különbségek vizsgálata
- 20. Single-cell RNA-seq: egyedi neuronok transzkriptomja, új sejttípusok felfedezése (pl. SOM, VIP alcsoportok)
- 21. Spatial transcriptomics: lokális génexpressziós térképek agyszeletekben, sejttípus, réteg és funkcionális környezet összekapcsolása
- 22. ATAC-seq, single-cell ATAC-seq: kromatin hozzáférhetősége: mely gének „nyitottak”, epigenetikai állapotok stresszre, tanulásra, drogokra
- 23. CUT&RUN / CUT&Tag: transzkripciós faktorok és epigenetikai markerek kötődése, sokkal érzékenyebb, mint klasszikus ChIP-seq
- 24. ChIP-seq: fehérje–DNS kölcsönhatások feltárása, fejlődési faktorok, CREB, MEF2, c-Fos stb.
- 25. Proteomika: tömegspektrometriás fehérjekimutatás: foszforilációs térképek, szinaptikus proteomika
- 26. Lipidomika, metabolomika: membránösszetétel, neurolipidek, glia–neuron interakciók metabolikus profilja
VI. Molekuláris anatómiai és sejttérképezési technikák: Kimondottan a molekulák térbeli elhelyezkedésének vizsgálatára, 2D és 3D szövettérképezésre és elemzésre fejlesztett módszerek. A mérést követő számítógépes megjelenítés során számtalan faktor kölcsönös előfordulását lehet velük megvizsgálni.
- 27. RNAscope: ultraspecifikus RNS in situ hibridizáció, ritka transzkriptumok kimutatása egyetlen sejten belül
- 28. CLARITY, iDISCO, uDISCO: átlátszóvá tett agyak, 3D immunfestés és génexpresszió vizsgálat
- 29. Multiplex immunhisztokémia: több tucat fehérje vizsgálata egyetlen agyszeleten, sejttípusok és szinaptikus mintázatok azonosítása
- 30. BARseq, MAPseq: molekuláris vonalkóddal ellátott projekciós térképezés
axonális célpontok DNS-alapú kódolása
VII. Molekuláris manipuláció és sejt-sors befolyásolása: Idegsejt és agyfejlődést vizsgáló eljárások. Hogyan fejlődnek a sejtvonalak és alakulnak ki az egyes agyterületek.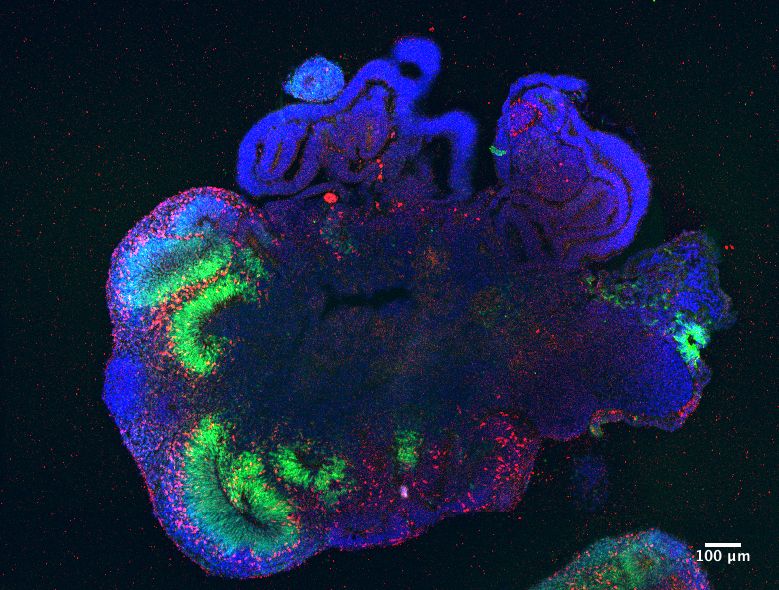
- 31. Lineage tracing (sejtvonal-követés): fejlődés során merre vándorolnak a neuronok, CRISPR-vonalkódolás (homing CRISPR barcoding) modern változatban
- 32. iPSC technológia: humán bőrsejtekből idegsejtek, asztrociták, oligodendrociták előállítása, betegspecifikus modellek
- 33. Organoidok (mini-agyak): 3D agyi szövetmodellek, fejlődés, betegségek, droghatások vizsgálata
- 34. In utero elektroporáció: fejlődő egérmagzat agyába juttatott DNS, réteg- és sejttípus-specifikus expresszió
VIII. Nukleinsav- és fehérjeinterakciók feltárása: A genetkai szabályozó hálózatok vizsgálhatók.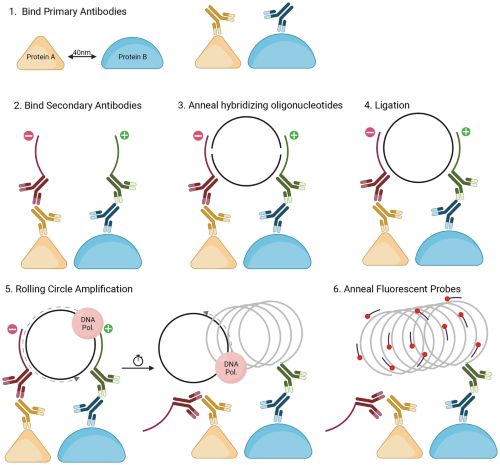
- 35. RNA-bindig protein mapping (CLIP-seq, HITS-CLIP, iCLIP), mely fehérjék milyen RNS-t kötnek, poszttranszkripciós szabályozás
- 36. Proximity ligation assays (BioID, APEX): fehérje–fehérje és fehérje–RNS közelítő interakciók térképei, sejten belüli mikrodomének feltárása (axon, dendrit, spine)
- 37. Ribosome profiling (Ribo-seq): mely mRNS-eket fordít le a riboszóma → valódi fehérjetermelés
IX. Optogenetikai módszerek és optikai szenzorok: Transzgénikus technológiákkal előállított feszültség, Ca, inegületátvivő anyag, metabolikus állapot, lipidanyagcsere-érzékelő molekulák, illetve serkentő és gátló opszinok. Ezek kombinációjával egyszerre lehet mérni és befolyásolni az agyi degsejt jelintegrációját és a hálózatokműködését. Illetve vizsgálni az ingerüetátvjvő anyagok felszabadulását és a sejtek anyagcsere állapotát. Az utóbbiakró hamarosan részletesen írunk.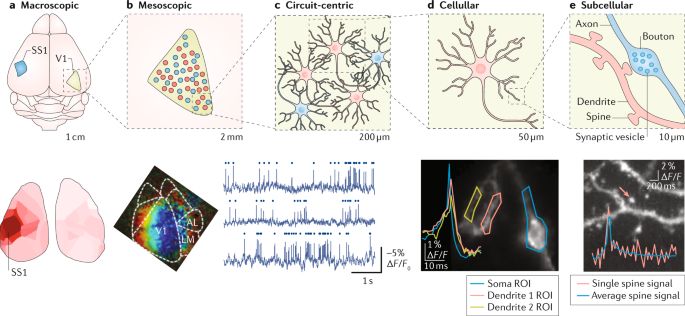
- 38. Kódolt Ca²⁺-szenzorok (GCaMP-verziók): neuronpopulációk működésének optikai követése
- 39. Kódolt feszültségszenzorok (GEVI-k): dendritikus integráció és AP-dinamika vizsgálata
- 40. All-optical physiology: optogenetika + Ca/voltage imaging kombinációja stimuláció és mérés egyszerre
X. Rendszerbiológia (Systems Biology) és Extrém modern multiomics: A rendszerbiológia az élő rendszereket komplex hálózatokként vizsgálja. Nem egyetlen génre vagy fehérjére fókuszál, hanem arra, hogyan hatnak egymásra: génhálózatok, jelátviteli utak, sejt-sejt kommunikációs csatornák, anyagcserefolyamatok.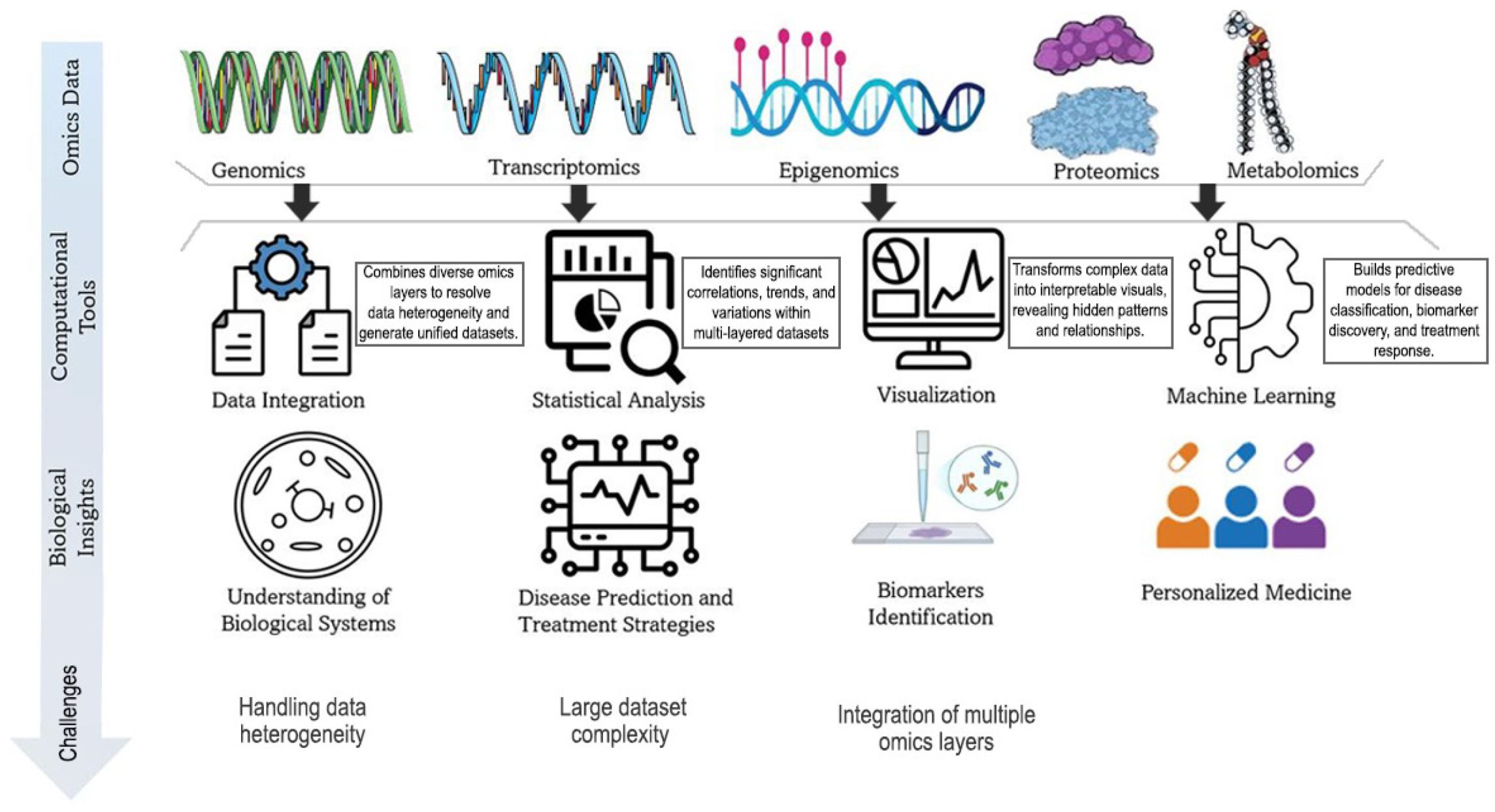 Az alapelv: a rendszer több, mint az alkotórészek összege. Az idegtudományban ez azt jelenti, hogy például a stressz, a tanulás, vagy a drogok hatásait nem egy-egy molekula szintjén vizsgáljuk, hanem az egész sejtprogram és hálózat változásait elemezzük.
Az alapelv: a rendszer több, mint az alkotórészek összege. Az idegtudományban ez azt jelenti, hogy például a stressz, a tanulás, vagy a drogok hatásait nem egy-egy molekula szintjén vizsgáljuk, hanem az egész sejtprogram és hálózat változásait elemezzük.
Itt számos nagy áteresztőképességű módszert kombinálnak, hogy egyetlen sejtben nézzék a szabályozó rendszerek összefüggéseit vagy egyes gének működésének megzavarását követő tovább gyűrűző változásokat figyelve szabályozó (jelátviteli, genetikus)- és anyagcsere hálózatokat derítsenek fel.
- 41. Multiomics single-cell megközelítések: scRNA-seq + scATAC-seq kombináció
génexpresszió + epigenetika egyetlen sejten - 42. Spatial multiomics: térbeli RNS, fehérje és epigenetikai rétegek összeolvasztása, agyszelet „molekuláris térképe”
- 43. CRISPR interference (CRISPRi) és CRISPR activation (CRISPRa): géncsendesítés vagy aktiválás DNS-vágás nélkül, stabil, finomhangolt génszabályozás neuronokban
- 44. Perturb-seq: CRISPR-alapú génzavarás + single-cell RNA-seq, funkcionális genomikai hálózatok feltárása
- 45. Mosaic analysis (MADM, MARCM): sejtspecifikus genetikai mozaikok létrehozása, fejlődés és funkció összekapcsolása
És ígérem itt az ámokfutás vége. Kiemelünk jónéhány sziporkázó technikát és elmerülünk részleteikben!
Szerző: Gulyás Attila






Köszönöm az Ünnepi jókivánságokat! Mint írtam ez valóban egy ámokfutás volt. A következő hezekben megjeenő blogbejegyzésekben érthetően és pánikot nem okozó formban körbejárjuk a módszerek zömét. Kérek eg kis kitartást addig is.