Újkeletű molekuláris-genetikai mérő módszerek: Ezt most mind mutasd meg, de gyorsan!
És akkor most lassú nyugalmas tempóban vágjunk bele az utóbbi években kidolgozott mérési és kísérleti módszerekbe. Megnézzük részleteikben hogyan működnek és mire használhatók.
Multiplex immunhisztokémia
Ez szigorúan véve nem genetikai technika, hiszen nukleinsav manipulációt nem alkalmaz. 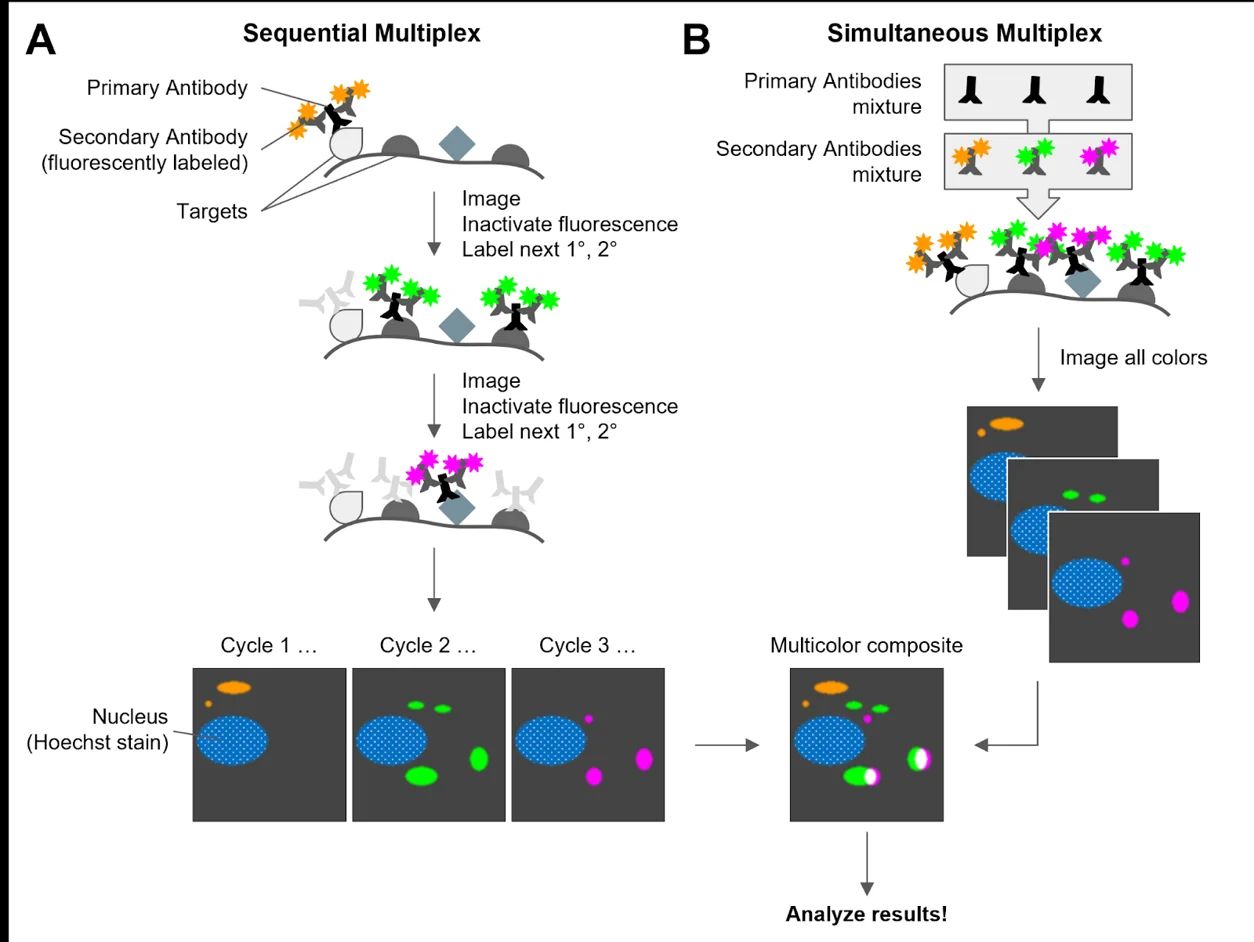 De, mint jelentős, 2025ben új módszert, itt érdemes bemutatni. A többszörös fluoreszens immunfestés extrém felturbózott alakja, alapelvei is hasonlóak.
De, mint jelentős, 2025ben új módszert, itt érdemes bemutatni. A többszörös fluoreszens immunfestés extrém felturbózott alakja, alapelvei is hasonlóak.
Nem egyetlen fehérjét, hanem 5–20 (új technikáknál akár több tucat) molekula elhelyezkedését vizsgáljuk ugyanazon a szövetszelvényen.
Két fő stratégia létezik: (1) Spektrális multiplexelés - Minden ellenanyag különböző színű festéket (fluorofórt) kap. A mikroszkóp spektrális felbontással külön választja a jeleket. 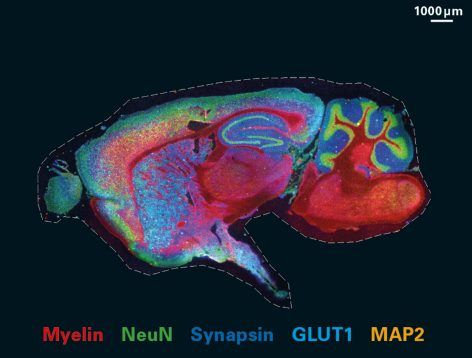 (2) Ciklikus eljárások - Egy körben 2–4 marker festése → kép készítése, majd fluorofór inaktiválása/elvonása. Újabb 2–4 marker festése → kép készítése. Végül a képeket algoritmus rendezi egyetlen multiplex képpé.
(2) Ciklikus eljárások - Egy körben 2–4 marker festése → kép készítése, majd fluorofór inaktiválása/elvonása. Újabb 2–4 marker festése → kép készítése. Végül a képeket algoritmus rendezi egyetlen multiplex képpé.
A módszer különösen hasznos az agykutatásban, mert megmutatja, hogyan helyezkednek el egymáshoz képest a különböző neuronok, gliasejtek vagy szinaptikus fehérjék, illetve hogyan változik a sejtek összetétele betegségekben. A multiplex immunhisztokémia tehát egy olyan „többrétegű szövettérkép”, amely egyszerre mutatja meg a szövet szerkezetét és molekuláris sokszínűségét.
Spatial transcriptomics: Hogyan lesz térkép a a gének kifejeződéséből.
A spatial (térbeli) transcriptomics lényege, hogy nemcsak azt tudjuk meg, milyen gének aktívak egy szövetben (milyen mRNS-eket találunk), hanem azt is, hogy hol aktívak. Ez azért forradalmi, mert a rétegekből, magokból és területekből álló agyban a génexpresszió sejtekre és rétegkre jellemző mintázatai sokszor csak térbeli elhelyezkedésüket figyelembe véve érthetők meg igazán. Megint előrángatva a szociológusokat. Annál az adatnál, hogy milyen a fizetések és az iskolai végzettségek eloszlása, sokkal többet mond, ha felrajzoljuk, hogy a különböző végzettségű és keresetű emberek, hol laknak Budapesten. Ebből például már megtudhatjuk, mely városrészekben milyen típusú feladatokat kell megoldani.
Ehhez nem elég tudni azt, hogy egy agyterületen ilyen és ilyen molekulák találhatók a sejtekben, hanem azt is kell látni, hogy a terület mely részének (mag, réteg), mely sejtjeiben, milyen molekulák fejeződnek ki együtt. A technika trükkje egyszerű elv köré épül. Minden mRNS-t „megjelölünk” a keletkezési helye alapján, majd ez a jelölés végig „rajta marad” a feldolgozás során.
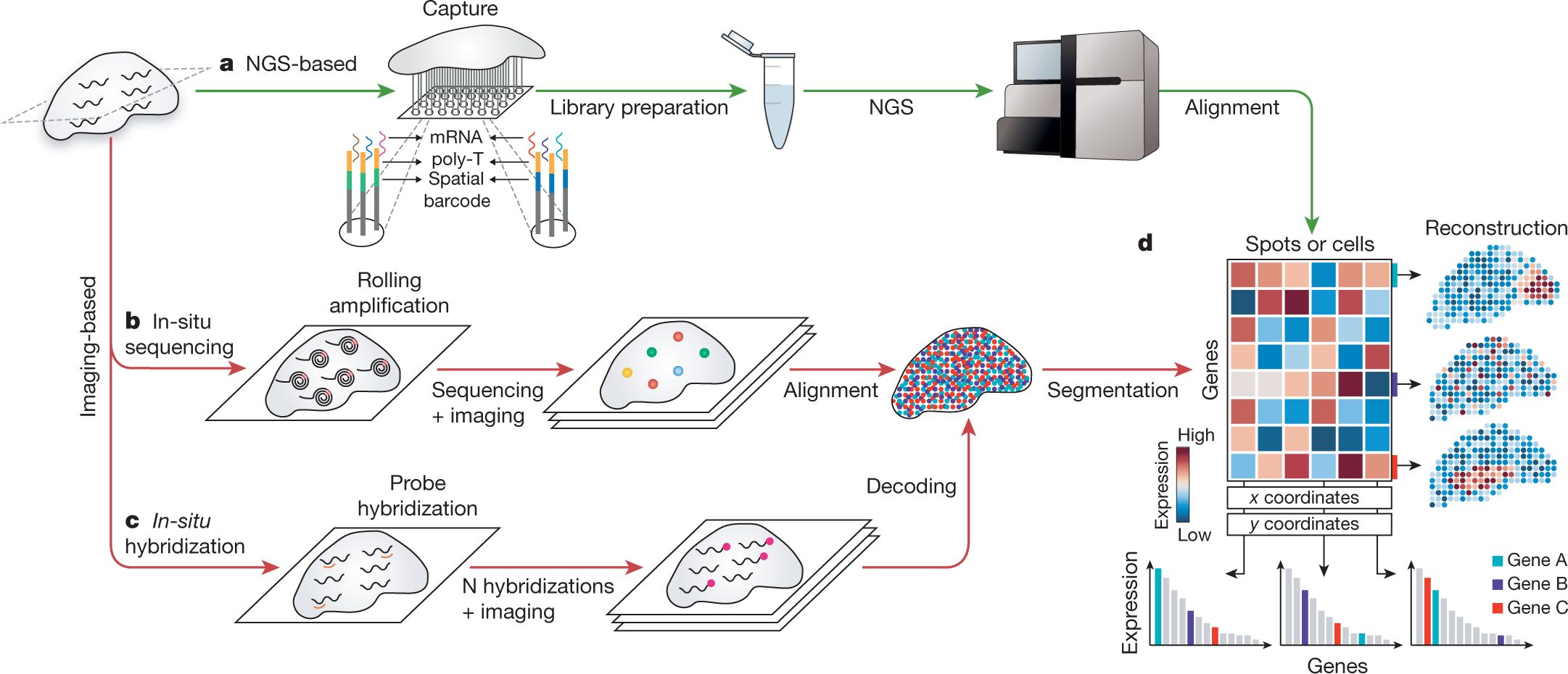
1. Lépés: Az agyszövetet gyorsan lefagyasztják (hogy az mRNS-eket megőrizzék), majd 5–10 µm vastag szeleteket vágnak. Ezeket ráhelyezik egy speciális tárgylemezre, amelyre korábban apró, láthatatlan RNS pozíciókód szekvenciákat nyomtattak („boltban” lehet ilyen lemezeket vásárolni). Ezek a mikroszkopikus kódmezők a térbeli információ kulcsa.
2. Lépés: A tárgylemez felülete mikroszkopikus rács, amelynek minden pontja tartalmaz egy egyedi térbeli vonalkód szekvenciát (barcode) és egy hozzá kapcsolt oligo(dT) szakaszt. Ez köti majd meg az mRNSek poli-A farkát. Ez a rács valójában több ezer minilabor, minden egyes pont egyedi azonosítót hordoz. Mintha a szövetet egy olyan papírra tennénk, amely négyzetrácsos, és minden négyzet más-más QR-kódot tartalmaz.
3. Lépés: Az mRNS-ek „ráfolynak” a kódokra. A szövetet fixálják és átjárhatóvá teszik. Ez azt jelenti: a sejtek kinyílnak, az mRNS-ek kiszabadulnak, és belefolynak a tárgylemez alatti mikropontokba. A kiszabadult mRNS-ek ott kötődnek meg, ahol épp „leszálltak” – vagyis azon a pixelhelyen, amely alatt a sejt volt. Ezzel a térbeli információ át is adódott, minden mRNS megkapja annak a pixelnek a vonalkódját, amely alatt volt.
4. Lépés: Az mRNS-ekről cDNS készül, amelybe beépül a térbeli vonalkód is, tehát a cDNS két információt tartalmaz: MELYIK génhez tartozik (szekvenciából tudjuk) és hogy HONNAN származik a szövetben (barcode alapján). Ez a kettős jelölés teszi lehetővé a térképezést.
Itt egy kis magyarázó kitérő. Mi az a cDNS és a reverse transcription? A biológiai rendszerek legtöbbjében (kivéve néhány vírust) a genetikai információt az RNS-nél ellenállóbb DNS hordozza.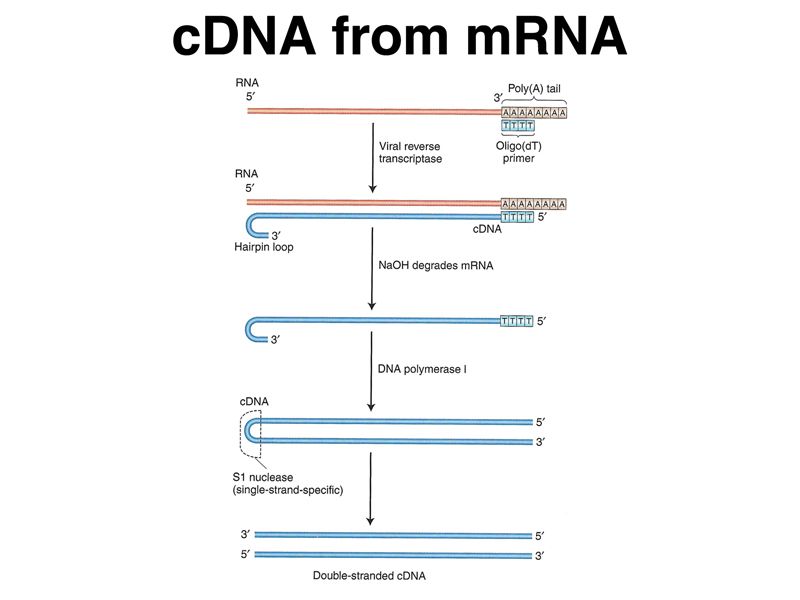 Erről íródnak át aztán a különböző RNS-ek, melyek vagy a fehérjeszintézishez szállítanak információt vagy pl. riboszóma alkatrészek lesznek. Ezért ugye a sejtekben a DNS replikálódik, az RNS nem. Tehát a sejtek enzimrendszere, amit mi is használunk a molekuláris biológiában, alapjában véve DNS manipulációra van „fejlesztve”. Ezért amikor RNS-információval szeretnénk dolgozni, annak információ tartalmát először átmásoljuk DNS-be, majd azzal dolgozunk. Az RNS -> DNS információ áramlási irány nem szokványos a biológiában, de néhány retrovírus rendelkezik olyan enzimekkel, amik ezt a másolást meg tudják csinálni, ez a reverse transcriptáz. A retrovírusok pontosan erről az akcióról kapták a nevüket. Alapjában véve RNS-t használó vírusok, de szaporodásukhoz először DNS-be másolják kódjukat, sőt ez beépül a genomba is, ahonnan évek múlva is újra előbújhatnak (ilyen az AIDS vírus is).
Erről íródnak át aztán a különböző RNS-ek, melyek vagy a fehérjeszintézishez szállítanak információt vagy pl. riboszóma alkatrészek lesznek. Ezért ugye a sejtekben a DNS replikálódik, az RNS nem. Tehát a sejtek enzimrendszere, amit mi is használunk a molekuláris biológiában, alapjában véve DNS manipulációra van „fejlesztve”. Ezért amikor RNS-információval szeretnénk dolgozni, annak információ tartalmát először átmásoljuk DNS-be, majd azzal dolgozunk. Az RNS -> DNS információ áramlási irány nem szokványos a biológiában, de néhány retrovírus rendelkezik olyan enzimekkel, amik ezt a másolást meg tudják csinálni, ez a reverse transcriptáz. A retrovírusok pontosan erről az akcióról kapták a nevüket. Alapjában véve RNS-t használó vírusok, de szaporodásukhoz először DNS-be másolják kódjukat, sőt ez beépül a genomba is, ahonnan évek múlva is újra előbújhatnak (ilyen az AIDS vírus is).
5. Lépés: A cDNS-ek összegyűjtése és szekvenálása (bázissorendjének, azaz információtartalmának meghatározása). A térbeli kódolt cDNS-eket leválasztják a tárgylemezről, majd nagymélységű szekvenálást végeznek (RNA-seq).
Az eredmény egy hatalmas lista:
Ez lényegében egy számítógépes puzzle, de minden darab (mRNS) megmondja, melyik négyzetből származik.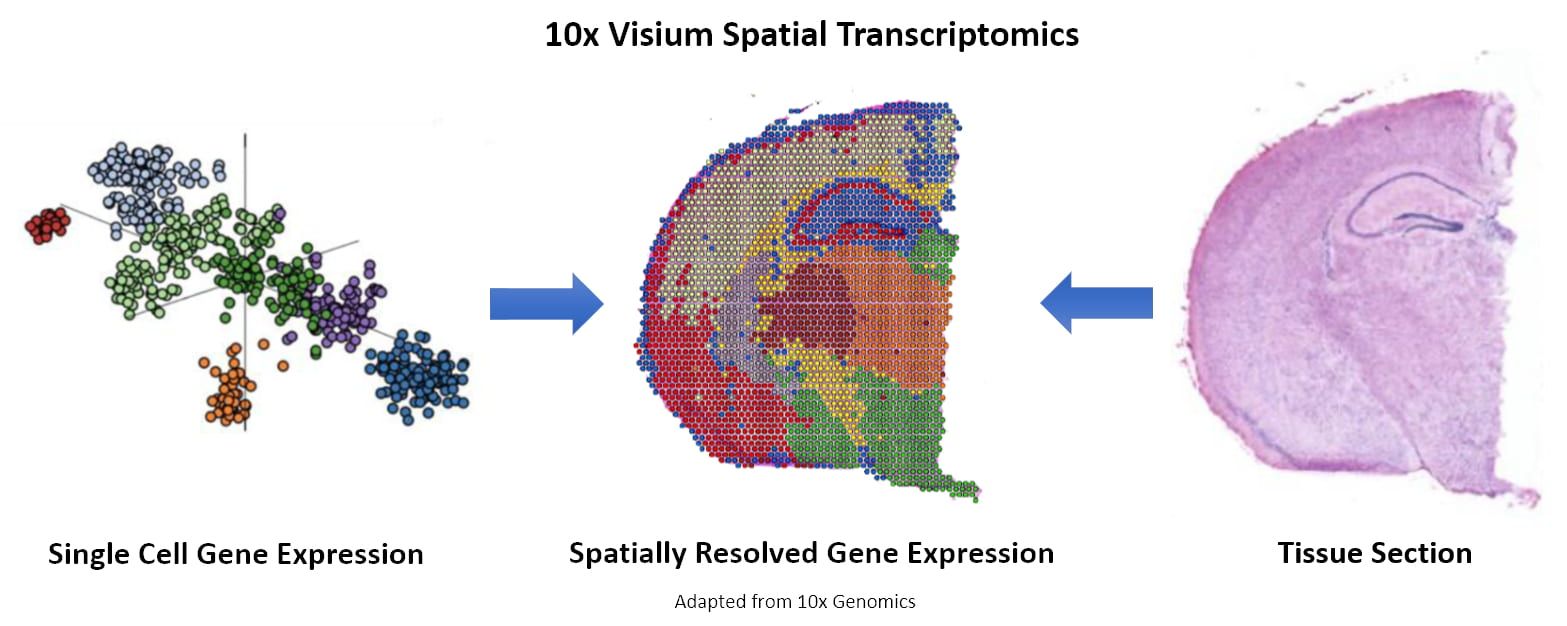
6. Lépés: Térképpé alakítás. A bioinformatika rekonstruálja a rács szerkezetét, melyik pixelben milyen mértékben fejeződött ki adott gén, hogyan különülnek el a sejttípusok térben, mely gének aktívak együtt egy-egy rétegben, magban, agyterületen. Az eredmény egy olyan hőtérkép, amely megmutatja, hol milyen gének „világítanak” a szövetben. Ez vizuálisan nagyon hasonlít az agyi aktivitást mutató fMRI térképekre – de itt nem véráramot, hanem konkrét gének működését látjuk. Ábrázolhatunk színekkel egyszerre több gént, de akár arányokat, összefüggéseket is megjeleníthetünk.
7. Lépés: Sejttípusok és anatómiai rétegek rekonstruálása.
Mivel egy pixelbe több sejt is eshet, a modern algoritmusok mért génkombinációk alapján sejttípusokat következtetnek és térbeli csoportosítást végeznek, a térkép rétegeit és határait automatikusan felismerik. Az eredményeket integrálják más adatbázisokkal (Allen Brain Atlas, Cell Types Database). Így megkapjuk mely rétegekben vannak pl. GABAerg sejtek, hol vannak serkentő projekciós neuronok, melyek a gliasejtek gyűjtőzónái, hogyan változik mindez betegség, fejlődés vagy stressz hatására.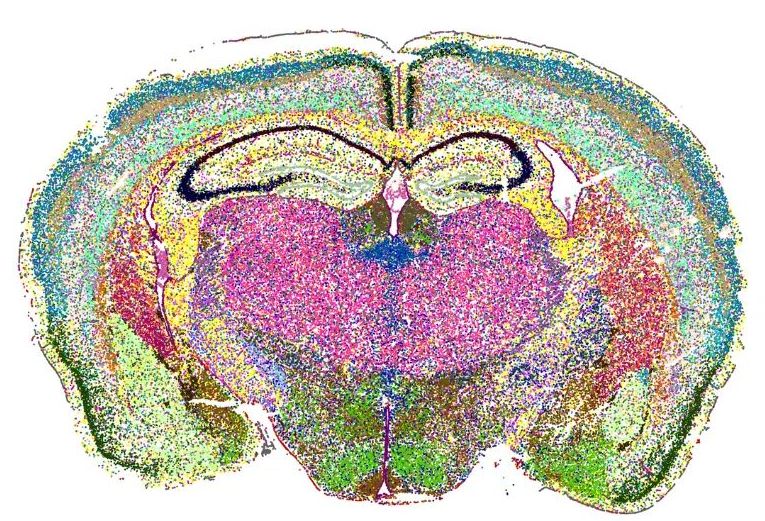
Röviden összefoglalva
A spatial transcriptomics kulcsa, hogy a szövetet egy kódolt rácsra helyezzük. Minden rácspont egyedi vonalkódot tartalmaz. A sejtekből „kiszabaduló” mRNS-ek megkapják ezt a kódot. A kód beépül a cDNS-be. A szekvenálás után minden molekuláról tudjuk, hogy melyik helyen volt. A génkifejeződés így térképpé válik.
Ez a módszer egyfajta molekuláris GPS az agyszövetben, megmutatja, hol vannak az egyes sejttípusok, milyen génhálózatok aktívak, és hogyan szerveződnek térben.
Nyilván ezt még lehet azzal barokkozni, hogy mondjuk az embrionális fejlődés során egyre későbbi időpontokban végezzük el ezt a térképezést, vagy például összehasonlítunk egy egészséges és egy beteg agyat. Így felderíthetjük a fejlődést mozgató térbeli kölcsönhatásokat vagy hogy egy-egy betegség mely sejtek változásából ered.
Szerző: Gulyás Attila





