MAPseq és BARseq, molekuláris projekció térképezés: ki-kivel van az agyban?
Emlékeztek még arra, a hőskorban hogyan néztük meg mely agyterület van összeköttetésben egy másikkal? Nyomkövető anyagokat, vagy kicsit később vírusokat adtunk be egy-egy agyterületre, majd immunfestéssel kimutattuk, hogy egyes sejtek hova küldik axonjaikat. Ezekkel a módszerekkel egyedi kísérletekkel lehetett felderíteni, hogy egy terület hova vetít (anterográd jelölés), vagy mely területek vetítenek egy helyre (retrográd jelölés). Az agyban azonban sok terület van, és a kombinatorikus robbanás miatt ezeket rengeteg lehetséges pálya kötheti össze. Arról nem is beszélve, hogy tapasztalatink szerint, egy agyterület különböző sejtcsoportjai más célterületre vetíthetnek. Szükség volt tehát olyan módszerekre, melyek egy menetben rengeteg vetítést derítenek fel, és még a kiinduló sejtek típusát is meghatározzák.
A bemutatandó két módszer kidolgozása erre az igényre adott megoldást. Mindkettő DNS-alapú módszer, amelyekkel axonpályákat lehet nagy felbontással követni, sok sejtre egyszerre.
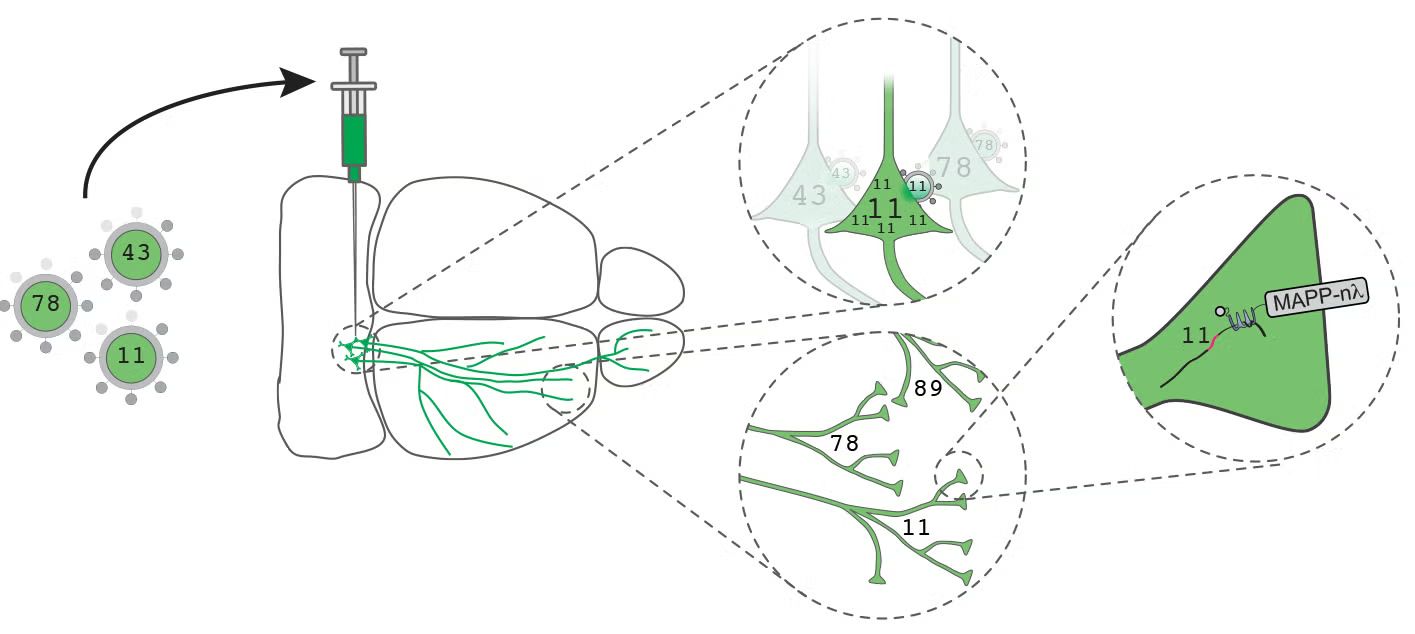
MAPseq (Multiplexed Analysis of Projections by sequencing): Vírusvektorral egyedi nukleinsav-barkódot (vonalkódot) juttatnak be sok idegsejtbe. Ennek hatására egyes idegsejtek axonágai ugyanazt a vonalkódot fejezik ki, mely megjeenik a célterületeken. A célterületek szövetmintáit felszeletelik, és szekvenálják az ott megjelenő vonalkódokat. Ha egy vonalkód X agyterületen jelenik meg, akkor a hozzá tartozó neuron oda vetít. Eredmény: több ezres projekciós mintázat egyetlen kísérletben.
BARseq (Barcoded Anatomy Resolved by sequencing): Fejlettebb változat, mely ugyanúgy vonalkódolt vírusokat ad be, de ezután térben lokalizálható in situ szekvenálást használ. Ez lehetővé teszi, hogy: megőrizzük a sejtek anatómiai helyét, és mégis több ezer kapcsolatot deríthessünk fel, így a teljes agy projekciós architektúrájának hatékony feltérképezésére alkalmas. Új sejttípusok – különösen vetítő neuronok – osztályozására, hálózati modellek ellenőrzésére (pl. PFC–talamusz–striátum kör finomszerkezetének felderítésére) használható.
A dolgok menete:
1. A kutatók előre elkészítenek egy vírusbankot (library-t), mely sok-sok (tízezres–százezres) különböző, véletlen nukleinsav-szekvenciát tartalmazó vírusrészecske-keveréke. Mindegyik barcode egy rövid (~30–100 bázis) random DNS-sor, így minden vírusrészecske egyedi kódot hordoz.
2. A víruskeveréket (AAV- vagy lentivírus) injekcióban egy adott agyterületre adják be (pl. V1, piriform cortex, hippokampusz CA1 stb.). A terület nagyságától és sejtszámától függően lehetőség szerint úgy hígítják a vírust, hogy egy sejtet átlagosan csak 1 virion fertőzzön meg.
3. A vírus bejut a neuronba, és kifejezi a barcode RNS-t. Ez az axonba is eljut, így a célterületeken lévő axonterminálisokban is kimutatható lesz a kód. Ezért tudják szekvenálni a célterületből az axonvetítések mintázatát. BARseq-ben a barcode DNS vagy RNS in situ kerül szekvenálásra, ezért térinformatikailag is megmarad a sejt és a vetítés helye és identitása.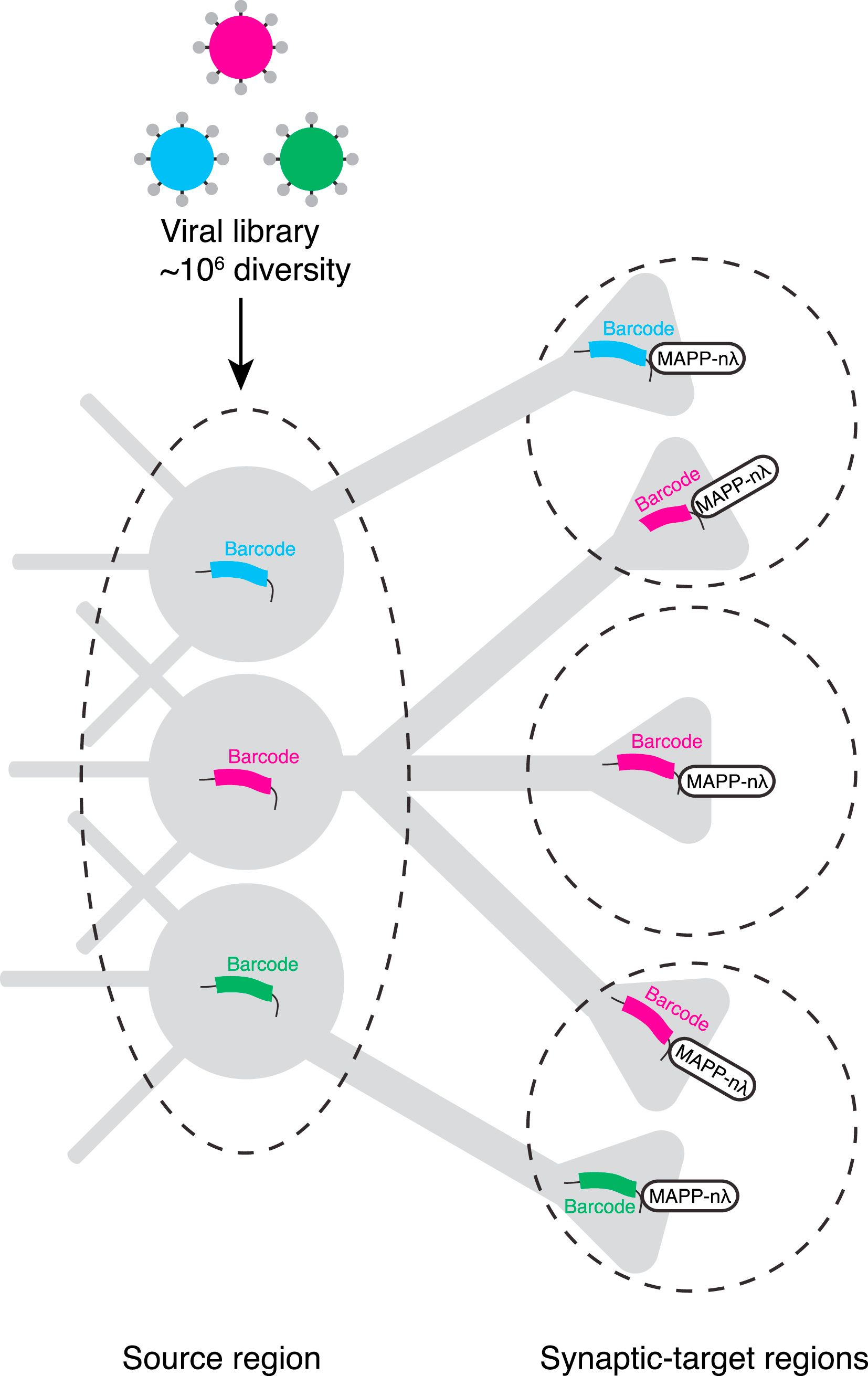
Számos kérdésre kaphatunk így választ:
1. Vajon ugyanabból az agyterületből a különböző sejtek eltérő célterületekre vetítenek-e? Példák: V1 vizuális kéreg rétegeinek sejtjei -> külön projekciós „típusok” (superior colliculus, lateralis geniculatus, másodlagos kéregmezők)
Piriform kéreg -> szétszórt, diffúz projekciók egyedi mintázatokkal
Prefrontális kéreg -> striatum és talamusz alterületeinek összeköttetése
2. Elágaznak-e a sejtek axonjai?
Ha egy sejt axonja több célterületre is vetít, akkor ugyanaz a molekuláris kód jelenik meg több különböző szöveti mintában. Ennek feltérképezése korábban rendkívül nehéz volt, mert hagyományos nyomkövető anyagokkal vagy teljesen töltötted a sejtet (de akkor egyetlen sejtről volt adat), vagy tömeges jelölést alapján kellett sejt-populációk vetítését átlagolni.
3. Mekkora az átfedés az egyes sejtek vetítése között?
A MAPseq/BARseq adatstruktúra természeténél fogva minden neuron egy vektor, amelynek dimenziói a célterületek, a vektor komponensei a barcode-felbukkanások aránya az adott területeken. Ez alapján mérhető két sejt mennyire vetít ugyanoda, mennyire különböznek a mintázatok, vannak-e jól elkülönült alcsoportok, gradiensek vagy folyamatos átmenetek? Az egyik nagy felismerés az volt, hogy sok agyterület nem homogén projekciós egység, hanem belül több sejtcsoport finoman eltérő vetítő mintázatot mutat. Ezt a hagyományos anatómia nem látta.
4. Sejttípusok összeköthetők projekciómintázatokkal. A BARseq in situ olvasása miatt, a sejt anatómiai helye, molekuláris profilja és projekciós mintázata összekapcsolható. Tehát megmondható például, hogy a felső V1 (elsődleges látókéreg) rétegek mely neuronjai vetítenek a superior colliculusba, és melyek a talamuszba, hogy piriform cortexben milyen molekuláris alcsoport milyen projekciós mintázattal jár, hogy a prefrontális kéreg mely alcsoportjai vetítenek a dorsalis vs. ventralis striatumba. Ez funkcionális-anatómiai „sejttípus × hálózat” mátrixot ad.
5. A teljes agyon belül térképezhető egyetlen axon jelenléte. MAPseq esetében bármely agyterület, amelybe az injektált populáció vetít, „felvillan”. Akár távoli vagy nagyon gyenge projekciókat is megtalál. Ez sokszor olyan célterületeket is kimutat, amelyeket klasszikus pályakövető technikák nem láttak vagy vitatottak voltak a gyenge jel miatt.
6. Nagyszámú sejt vetítésének vizsgálata statisztikai alapú hálózati elemzést tesz lehetővé. Projekciós csoportokat, funkcionális modulokat, hálózati hierarchiákat (pl. PFC -> talamusz -> striatum rendszer) lehet precízen feltérképezni. Ez alapvető változást hozott a „connectomics” területére, mert sejtszinten, nem csupán rostkötegekkel dolgozik. Radásul a tömeges connectome eredményeket nyújtó DTI-MRI-vel ellentétben a vetítések irányáról is beszámol.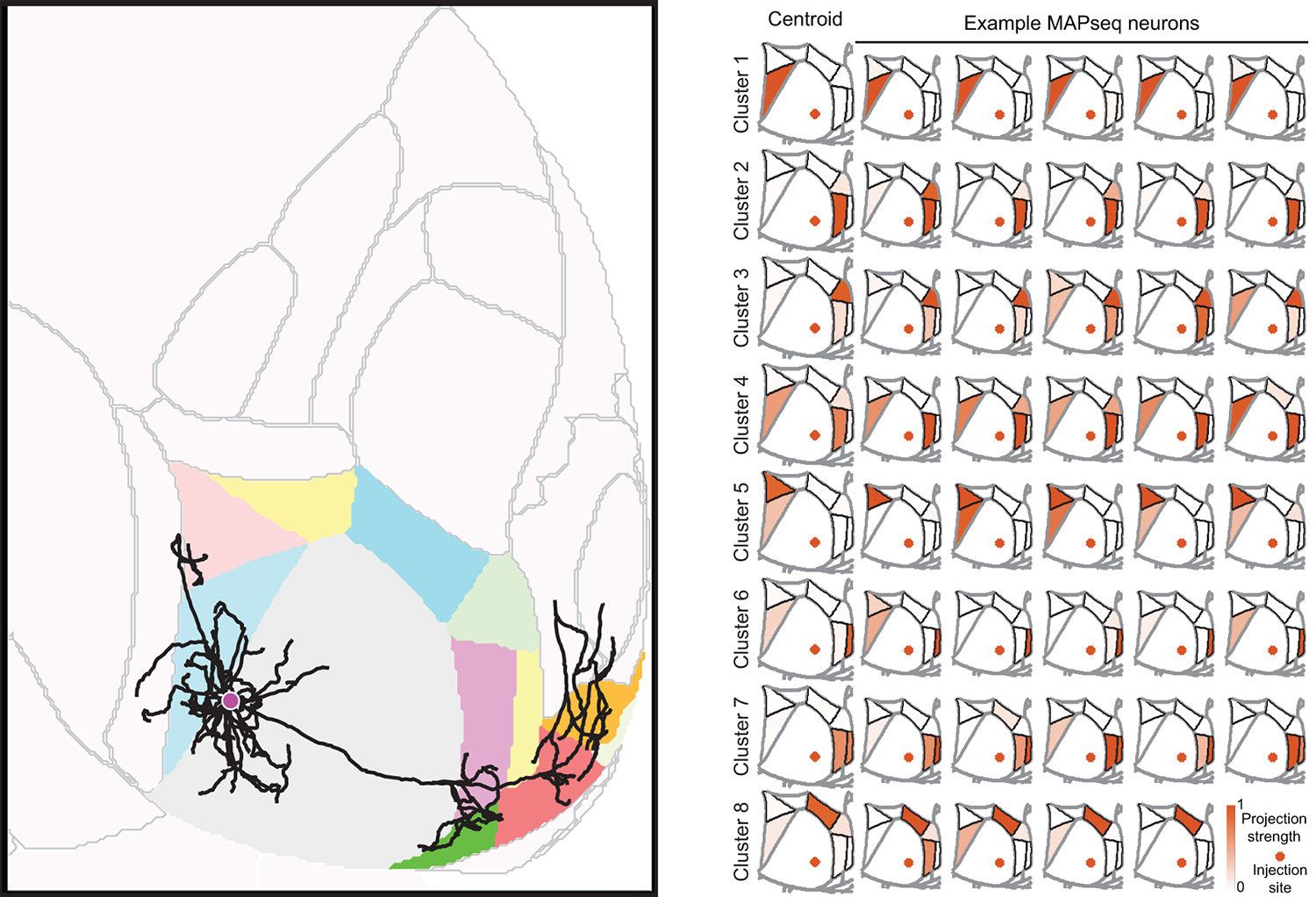
A MAPseq és BARseq segítségével tehát meg lehet mondani:
✔ hogy egy régió különböző neuronjai hova vetítenek
✔ hogy egy axon elágazik-e és hány célterülete van
✔ hogy mennyire fedik át egymást a neuronok projekciós célpontjai
✔ hogy a projekciók erőssége milyen mintázatot követ
✔ hogy a projekciók összekapcsolhatók-e molekuláris sejttípusokkal
✔ hogy teljes agyon belül „hol bukkan fel” egy adott neuron axonja
Tehát a módszerek nem csak azt mondják meg, hova megy egy axon, hanem a projekciós logika mögötti sejttípus- és hálózatszerveződést is feltárják.
A következőkben a hasonlóan nagy áteresztőképességű „omics” technikákkal folytatjuk.
Szerző: Gulyás Attila





